AG Schüller (em.) - Hefephysiologie
Die AG Schüller behandelt thematisch die Themen der transkriptionalen Regulation von Strukturgenen der Phospholipidbiosynthese und die kohlenstoffquellenabhängige Expression gluconeogenetischer Strukturgene in der Hefe Saccharomyces cerevisiae.
Projekt „Genetische Kontrolle der Phospholipid-Biosynthese in der Bäckerhefe Saccharomyces cerevisiae“
Allgemeine Informationen:
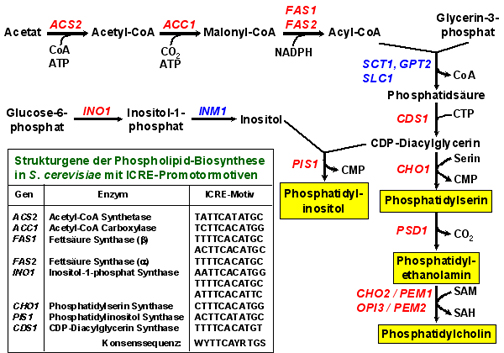
Strukturgene der Phospholipid-Biosynthese in der Hefe Saccharomyces cerevisiae werden durch die verfügbare Menge der Vorläufermoleküle Inositol und Cholin (IC) im Medium transkriptional reguliert (Übersicht). Diese Gene tragen zumindest eine Kopie der 11 bp langen Aktivierungssequenz ICRE (“inositol/choline-responsive element”), an die ein heterodimerer Komplex der Aktivatoren Ino2 und Ino4 bindet. Ino2 und Ino4 gehören zur Familie der basischen Helix-Loop-Helix-Proteine (bHLH), zu der auch die strukturverwandten humanen Protoonkoproteine c-Myc und Max zählen. Bei IC-Mangel rekrutiert Ino2 generelle Faktoren der Transkription (z. B. den basalen Transkriptionsfaktor TFIIB, den Mediator-Komplex, Chromatinremodellierungs-komplexe wie SWI/SNF und Ino80 sowie Histonacetyltransferasen wie Gcn5 im SAGA-Komplex) und steigert die Expression z. B. des Inositol-1-phosphat Synthase Gens INO1 und der Fettsäure Synthase Gene FAS1 und FAS2. Am deutlichsten zeigt sich die Genregulation beim Inositol-1-phosphat Synthase Gen INO1, das ca. 50-fach reprimiert wird, wenn IC umgekehrt in hoher Menge vorliegen. Unter diesen Bedingungen kontaktiert der Repressor Opi1 den Aktivator Ino2 und bewirkt eine Veränderung der Chromatinstruktur in den Promotorregionen der Zielgene. Hierzu bindet Opi1 seinerseits an sog. Corepressor-Komplexe wie Sin3 und Cyc8/Tup1, die schließlich Histondeacetylasen kontaktieren und ein lokal unzugängliches Chromatin etablieren.
Untersuchte Fragestellungen:
- Regulatorische Sequenzen vor Strukturgenen der Phospholipid-Biosynthese in Hefe
- Interaktion spezifischer Regulatorproteine der Phospholipid-Biosynthese (Aktivatorproteine Ino2 und Ino4, Repressorprotein Opi1) mit generellen Faktoren der transkriptioalen Aktivierung (Mediator-Komplex; basale Transkriptionsfaktoren, z. B. TFIIB = Sua7) oder Repression (Sin3-Corepressorkomplex, Cyc8/Tup1-Corepressorkomplex)
- Bedeutung pleiotroper Faktoren der Chromatinremodellierung (Swi2-Komplex, Ino80-Komplex) bzw. Chromatinmodifizierung (Histonacetyltransferasen HAT, z. B. SAGA-Komplex mit Gcn5; Histondeacetylasen HDAC, z. B. Rpd3, Hda1, Hos1; Histonmethyltransferasen HMT, z. B. Set2)
Weiterführende Informationen:
Alle im Text fett markierten Genabschnitte können unter der Suche von yeastgenome.org nachgeschlagen werden, um weitere Informationen zu erhalten.
Projekt „Genetik der Coenzym A Biosynthese in der Bäckerhefe Saccharomyces cerevisiae“
Allgemeine Informationen:
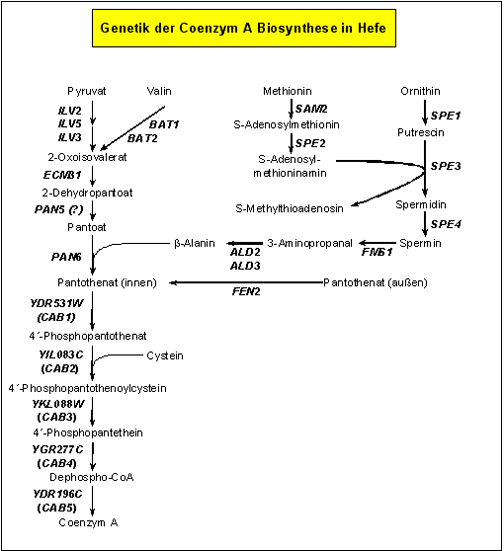
Coenzym A (CoA) dient in allen Organismen als Überträger von Acylgruppen, die als energiereiche Thioester mit unterschiedlichen Kettenlängen (v. a. als Acetyl-CoA, C2, bis hin zu Stearoyl-CoA, C18 und ggf. länger) in zahlreiche anabole und katabole Stoffwechselwege eingehen. Besonders Acetyl-CoA mit seinem hohen Gruppenübertragungspotenzial steht im Zentrum des metabolischen Geschehens und betrifft Prozesse wie die Fettsäure-Biosynthese und -Degradation, die Sterol-Biosynthese, die Gluconeogenese bzw. Glyoxylat-Zyklus aus C2-Substraten (v. a. in Mikroorganismen), zahlreiche Wege des Sekundärstoffwechsels (Synthese einiger Antibiotika) sowie Acetylierungsreaktionen von Alkoholen, Aminozuckern und v. a. Proteinen, wobei die Histon-Acetylierung in den letzten 15 Jahren als herausragende Modifikation im Chromatin eukaryotischer Zellen identifiziert wurde. Die Biosynthese von CoA selbst läßt sich in zwei Abschnitte unterteilen (Synthese von Pantothenat = Vitamin B5 aus Valin-Derivaten und beta-Alanin; Transfer von ATP und Cystein auf Pantothenat; siehe: Verlauf der CoA-Biosynthese in Hefe) und erfordert den Beitrag mehrerer Aminosäuren, wobei die vom Cystein stammende SH-Gruppe für die Reaktivität des Coenzyms von zentraler Bedeutung ist. Die Pantethein-Einheit des CoA wird auch für das reaktive Zentrum von Enzymen beispielsweise der Fettsäure- bzw. Polyketidsynthese verwendet (ACP, Acyl Carrier Protein).
Untersuchte Fragestellungen:
- Strukturgene der Pantothenat- und Coenzym A-Biosynthese in Hefe
- Allosterische Regulation der Pantothenat Kinase und Konstruktion deregulierter Enzymvarianten
- Interaktion zwischen Enzymen der Coenzym A-Biosynthese:
- Pantothenat Kinase (PanK; Gen: CAB1)
- 4'-Phosphopantothenoylcystein synthetase (PPCS; Gen: YIL083C, CAB2)
- 4'-Phosphopantothenoylcystein Decarboxylase (PPCDC; Gen: YKL088W, CAB3)
- 4'-Phosphopantethein Adenylyltransferase (PPAT; Gen: YGR277C, CAB4)
- Dephospho-CoA Kinase (DPCK; Gen: YDR196C, CAB5)
- Überexpression von Coenzym A Biosynthesegenen zur Erhöhung der zellulären CoA-Ausbeute
Weiterführende Informationen:
Cab1
www.yeastgenome.org