Projekte
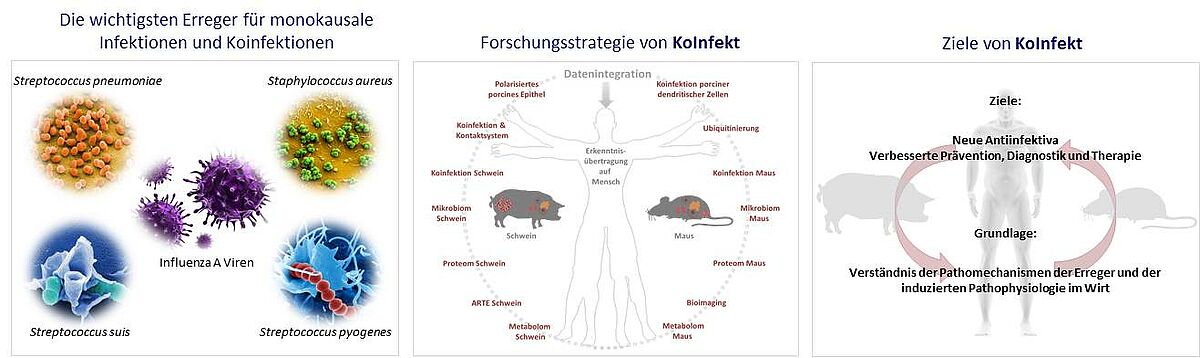
Das Projekt KoInfekt hat zum Ziel den Krankheitsverlauf, die Pathomechanismen und die Immunantwort des Wirtes im Kontext von bakto-viralen Koinfektionen in experimentellen in vitro und ex vivo Modellen mit Primärzellen sowie relevanten in vivo Erreger-Wirts-Systemen aufzuklären. Virale Infektionen im Nasen- und Rachenraum sind die Wegbereiter für bakterielle Sekundärinfektionen mit Pathobionten, die den Krankheitsverlauf entscheidend beeinflussen. Diese Koinfektionen, insbesondere die Koinfektionen von Influenza A Viren (IAV) mit Pneumonie-auslösenden Bakterien wie S. pneumoniae, S. aureus sowie S. pyogenes, stellen wegen der weltweit hohen Morbiditäts- und Mortalitätsraten ein großes Problem für die Gesundheitssysteme dar.
In KoInfekt soll die Ko-Pathogenese von IAV und den o.g. Pathobionten bezüglich synergistischer oder antagonistischer Effekte umfassend unter Einbeziehung von in vivo und ex vivo Bioimaging untersucht werden. Die induzierten Immunantworten, der Einfluss des Mikrobioms, Veränderungen im Stoffwechsel der infizierten Gewebe, Schädigungen der Epithelbarrieren und die Identifizierung molekularer Biomarker durch detaillierte Proteomanalysen sind zentrale Bestandteile des Forschungsprogramms. Die in vivo Studien erfolgen im relevanten natürlichen Erreger-Wirts-System Schwein vergleichend zu experimentellen Infektionsmodellen der Maus. Das Ziel von KoInfekt, das Schwein als experimentell zugängliches, biomedizinisches Infektionssystem für die bakto-viralen Koinfektionen zu etablieren, ist dabei von herausragender Bedeutung für zukünftige Studien mit humanpathogenen Erregern bei monokausalen bzw. Koinfektionen. Mit dem Schwein soll ein dem Menschen vergleichbares Infektionsmodell etabliert werden.
Das Gesamtkonzept von KoInfekt soll die Basis für eine schnellere und effizientere Translation der in Infektionsstudien erzielten Ergebnisse auf den Menschen bilden. Die Ergebnisse aus KoInfekt und weiteren Studien im Schwein sollen neue Strategien für die Bekämpfung und Prävention monokausaler und Koinfektionen für die klinische Praxis aufzeigen.
Folgende wissenschaftliche Fragestellungen bzw. Teilprojekte werden in KoInfekt bearbeitet (in alphabetischer Reihenfolge):
Aufgrund der weiten Verbreitung von Antibiotikaresistenzen sind alternative therapeutische Interventionen notwendig. Konventionelle Ziele für die Therapie mit Antisense-Peptidnukleinsäuren (Antisense-Peptide-Nucleic-Acids, PNAs) sind die virale Replikation und das bakterielle Wachstum. Darüber hinaus werden in einem innovativen Ansatz multiresistente Keime durch die Anwendung von Antisense-PNAs gegen bestehende Antibiotika-Resistenzen resensibilisiert. Zunächst werden zellpenetrierende Antisense-PNAs in vitro und ex vivo auf ihre Funktionalität geprüft und anschließend erfolgt die Wirksamkeitsprüfung einer PNA Therapie unter in vivo Bedingungen.
ProjektleiterInnen:
Prof. Dr. Bernd Kreikemeyer (Universitätsmedizin Rostock)
Projektmitarbeitende:
Postdoktorandin:
Dr. Sonja Oehmcke-Hecht (Universitätsmedizin Rostock)
Doktorandin:
Gina Barkowsky (Universitätsmedizin Rostock)
Die pathophysiologischen Effekte, die Immunantwort sowie das Metabolom und (Immuno-)Proteom sollen im Kontext der Koinfektionen in der Maus analysiert werden und mit den Ergebnissen aus den monokausalen Infektionen und Koinfektionen im Schwein verglichen werden. Dabei soll auch zwischen leichten und schweren Krankheitsverläufen unterschieden sowie die Altersabhängigkeit der Pathogenese und Immunantwort bei Koinfektion untersucht werden. Zusätzlich wird untersucht, ob die Koinfektion und damit assoziierte verschlimmerte bakterielle Lungenentzündung (Pneumonie) die Häufigkeit und Schwere einer bakteriellen Gelenkentzündung (Arthritis) erhöht. Die Ergebnisse und Analysen sollen nachfolgend auf das Schwein übertragen werden können.
ProjektleiterInnen:
Prof. Dr. Sven Hammerschmidt (Universität Greifswald)
Prof. Dr. Bernd Kreikemeyer (Universitätsmedizin Rostock)
Prof. Dr. Brigitte Müller-Hilke (Universitätsmedizin Rostock)
Projektmitarbeitende:
NachwuchsgruppenleiterInnen:
Dr. Nikolai Siemens (Universität Greifswald)
Dr. Sonja Oehmcke-Hecht (Universitätsmedizin Rostock)
DoktorandInnen:
Fabian Cuypers (Universität Greifswald)
NN (Universität Greifswald)
Gina Barkowsky (Universitätsmedizin Rostock)
Johann Volzke (Universitätsmedizin Rostock)
Das Schwein soll als biomedizinisches Modell für Koinfektionen, klinische Messparameter und Testsysteme etabliert werden. Nach der Etablierung standardisierter Infektionsmodelle monokausaler und Koinfektionen mit geeigneten Schweinezuchtlinien und Pathogenen, Influenza und S. suis, werden klinische, immunologische, infektiologische und pathologische Parameter nach monokausalen bzw. Koinfektionen analysiert. Bedeutsam ist, dass die etablierten Infektionsmodelle die Untersuchung von a) veterinärmedizinisch bedeutsamen Koinfektionen im Schwein, b) Koinfektionen im Schwein mit hohem zoonotischem Potential und c) Koinfektionen beim Menschen im biomedizinischen Modell Schwein erlauben.
Projektleiter:
Prof. Dr. Dr. h.c. Thomas C. Mettenleiter (FLI, Riems)
Projektmitarbeitende:
Nachwuchsgruppenleiterin:
Dr. Theresa Schwaiger (FLI, Riems)
DoktorandInnen:
Alexander Schäfer (FLI, Riems)
Jean-Pascal Hoffmann (FLI, Riems)
Da das Schwein als biomedizinisches Infektionsmodell etabliert werden soll, ist dessen Immunantwort gegen Viren, Bakterien, einzelne Antigene oder polyvalente Impfstoffe von zentralem Interesse. Deshalb wird die adaptive Immunantwort, primär die Antikörperantwort, nach Immunisierung mit verschiedenen bakteriellen Antigenen im Vergleich zur humanen Immunantwort analysiert sowie der Einfluss der Immunisierung auf die Influenzainfektion und umgekehrt untersucht. Dabei sollen auch die grundlegenden Fragen beantwortet werden, gegen wie viele Antigene das adaptive Immunsystem simultan eine starke Antwort aufbauen kann und wie sich eine vorhergehende Impfung auf die Immunabwehr gegen IAV-Infektion auswirkt.
ProjektleiterInnen:
Prof. Dr. Barbara M. Bröker (Universitätsmedizin Greifswald)
Prof. Dr. Uwe Völker (Universitätsmedizin Greifswald)
Prof. Dr. Dr. h.c. Thomas C. Mettenleiter (FLI, Riems)
Projektmitarbeitende:
Nachwuchsgruppenleiterin:
Dr. Theresa Schwaiger (FLI, Riems)
Postdoktorand:
Dr. Vincent Péton (Universitätsmedizin Greifswald)
Doktoranden:
Christoph Decker (Universitätsmedizin Greifswald)
Alexander Schäfer (FLI, Riems)
Jean-Pascal Hoffmann (FLI, Riems)
CD4-positive T-Zellen sind für die Entwicklung einer schützenden Immunantwort entscheidend, können aber auch immunpathogene Prozesse antreiben. Nach Anreicherung von antigen-spezifischen CD4+CD154+ T-Zellen mit Hilfe der ARTE (Antigen-reaktive T-Zell Anreicherung)-Technik sollen diese vor und nach Koinfektion auf funktioneller und molekularer Ebene charakterisiert, und die regulatorischen Funktionen der CD4+CD154+ Zellen untersucht werden.
ProjektleiterInnen:
Prof. Dr. Barbara M. Bröker (Universitätsmedizin Greifswald)
Prof. Dr. Dr. h.c. Thomas C. Mettenleiter (FLI, Riems)
Projektmitarbeitende:
NachwuchsgruppenleiterInnen:
Dr. Theresa Schwaiger (FLI, Riems)
Postdoktorand:
Dr. Vincent Péton (Universitätsmedizin Greifswald)
Doktoranden:
Alexander Schäfer (FLI, Riems)
Jean-Pascal Hoffmann (FLI, Riems)
Ziel ist die Untersuchung des Effekts von Immunisierungen und Infektionen/Koinfektionen auf die Zusammensetzung von Plasma- bzw. Gewebeproteomen (Lunge und Bronchoalveolarlavage) und auf das Antikörperprofil in Schwein und Maus. Zunächst werden die Basisproteome und Basisantikörperprofile bzw. deren Variation in nicht-infizierten und nicht-immunisierten Tieren als Basislinie aufgenommen, um sie dann im weiteren Verlauf des Projektes zur Berechnung quantitativer Unterschiede nach Infektionen/Koinfektionen und Immunisierung mit viralen bzw. bakteriellen Antigenen zu verwenden.
ProjektleiterInnen:
Prof. Dr. Barbara M. Bröker (Universitätsmedizin Greifswald)
Prof. Dr. Uwe Völker (Universitätsmedizin Greifswald)
Prof. Dr. Sven Hammerschmidt (Universität Greifswald)
Projektmitarbeitende:
Nachwuchsgruppenleiter:
Dr. Nikolai Siemens (Universität Greifswald)
PostdoktorandInnen:
Dr. Manuela Gesell Salazar (Universitätsmedizin Greifswald)
Dr. Vincent Péton (Universitätsmedizin Greifswald)
Doktorand:
Christoph Decker (Universitätsmedizin Greifswald)
Ziel ist die Aufklärung der Zusammensetzung des Lungen-Mikrobioms von nicht-infizierten, monokausal- und koinfizierten Schweinen bzw. Mäusen und der systemischen Effekte der respiratorischen Infektionen auf das Darm-Mikrobiom. Dazu werden die phylogenetische Zusammensetzung der Lungen- und Darm-Mikrobiome und deren Expressionsprofile mit Hilfe von 16S rRNA Gen- und Meta-Omics Analysen bestimmt und vergleichend charakterisiert.
ProjektleiterInnen:
Prof. Dr. Dr. h.c. Thomas C. Mettenleiter (FLI, Riems)
Prof. Dr. Katharina Riedel (Universität Greifswald)
Prof. Dr. Tim Urich (Universität Greifswald)
Projektmitarbeitende:
Nachwuchsgruppenleiterin:
Dr. Theresa Schwaiger (FLI, Riems)
DoktorandInnen:
Claudia Karte (FLI, Riems)
Laurin Christopher Gierse (Universität Greifswald)
Alexander Meene (Universität Greifswald)
Infektionen sind mit einer metabolischen Adaptation assoziiert, die Erreger-spezifisch ausfallen kann. Ziel ist die Untersuchung gelöster Metabolite und volatilen organischen Verbindungen (VOCs) aus Körperflüssigkeiten und Gewebeextrakten von nicht-infizierten und infizierten Tieren bzw. aus Infektionen in der Zellkultur. Außerdem soll eine Lipidomics-Plattform aufgebaut werden. Damit sollen die relevanten Metabolite des zellulären Lipidstoffwechsels abgedeckt und erstmals eine Analyse des infektionsrelevanten Lipid-Metabolismus ermöglicht werden.
ProjektleiterInnen:
Prof. Dr. Michael Lalk (Universität Greifswald)
Prof. Dr. Jochen Schubert (Universitätsmedizin Rostock)
Projektmitarbeitende:
Postdoktorandinnen:
Dr. Karen Methling (Universität Greifswald)
N.N. (Universitätsmedizin Rostock)
Mit Dimensionsreduktionsverfahren und regularisierten maschinellen Lernverfahren erfolgt eine Konzentrierung und Reduktion der Daten, um diese einer integrativen Analyse zugänglich zu machen. Anschließend werden die Daten der verschiedenen experimentellen Plattformen auf molekulare Netzwerke abgebildet, um von den reinen Dateninhalten hin zu funktionellen Analysen mit dem Ziel der Interpretierbarkeit der biologischen und medizinischen Verhaltensweisen zu gelangen. Mittels Netzwerkinferenzverfahren und mathematischer Modellierung werden zentrale Mechanismen der Wirts-Pathogen-Interaktionen und der Wirtsantwort auf Infektionen und Koinfektionen charakterisiert, um neue, experimentell testbare Hypothesen zu zentralen Mechanismen in silico zu generieren und zu testen.
ProjektleiterInnen:
Prof. Dr. Lars Kaderali (Universitätsmedizin Greifswald)
Projektmitarbeitende:
Postdoktorand:
Dr. Anriban Bhar
Doktorandin:
Dana Kleimeier
Das Kontaktsystem (KS), Teil der angeborenen Immunität, besteht aus einer Gruppe von Plasmaproteinen, die an der Blutgerinnung beteiligt sind. Virale und bakterielle Erreger modulieren das KS und beeinflussen so den Krankheitsverlauf. Die Aktivierung des KS durch IAV bzw. durch die Pathobionten wird im Plasma (ex vivo) und nach monokausaler und Koinfektion vom Schwein bzw. Maus untersucht. In vivo soll durch Blockierung der identifizierten KS-Faktoren bzw. der Behandlung mit monoklonalen Antikörpern die Bedeutung für die Pathophysiologie der Infektion und die Nutzung als neuartige therapeutische Intervention analysiert werden.
ProjektleiterInnen:
Prof. Dr. Bernd Kreikemeyer (Universitätsmedizin Rostock)
Projektmitarbeitende:
Postdoktorandin:
Dr. Sonja Oehmcke-Hecht (Universitätsmedizin Rostock)
Doktorandin:
Gina Barkowsky (Universitätsmedizin Rostock)
Dendritische Zellen (DCs) sind wichtige professionelle Phagozyten und Antigen-präsentierende Zellen (APCs) des angeborenen Immunsystems. Nach Charakterisierung der porcinen DCs und Etablierung von monokausalen Infektionen und Koinfektionen humaner bzw. porciner DCs mit IAV und den bakteriellen Erregern S. suis, S. pneumoniae bzw. S. aureus werden die immunologischen Reaktionsmuster der DCs auf die Infektionen analysiert und verglichen. DCs verbinden das angeborene mit dem erworbenen Immunsystem. Daher werden in Kokultivierungsversuchen mit T-Zellen die Aktivierung der T-Zellen und ihre Zytokinexpression betrachtet. Die DC-Infektionen werden auch in der Fluoreszenzmikroskopie und Elektronenmikroskopie visualisiert.
ProjektleiterInnen:
Prof. Dr. Sven Hammerschmidt (Universität Greifswald)
Prof. Dr. Dr. h.c. Thomas C. Mettenleiter (FLI, Riems)
Prof. Dr. Barbara M. Bröker (Universitätsmedizin Greifswald)
Projektmitarbeitende:
NachwuchsgruppenleiterInnen:
Dr. Nikolai Siemens (Universität Greifswald)
Dr. Theresa Schwaiger (FLI, Riems)
Postdoktorand:
Dr. Vincent Péton (Universitätsmedizin Greifswald)
DoktorandInnen:
N.N. (Universität Greifswald)
Surabhi Surabhi (Universität Greifswald)
Alexander Schäfer (FLI, Riems)
Luca Zaeck (FLI, Riems)
Das Infektionsmodell Schwein bietet die Möglichkeit, in vivo Ergebnisse direkt mit in vitro Zellkulturmodellen vergleichend zu untersuchen. Die Kolonisierung, Invasion bzw. Transmigration und induzierten Wirtszellreaktionen bei monokausalen sowie Koinfektionen von primären porcinen Epithelzellen, u.a. im ALI („Air Liquid Interface“) System, mit IAV, S. suis oder S. pneumoniae werden analysiert. Für S. pneumoniae soll auch die Rolle von Virulenzfaktoren und zellulären Rezeptoren aufgezeigt werden. Außerdem werden die apikalen, basalen und intrazellulären Vorgänge am/im Epithel bei den Koinfektionen, auch aus infiziertem Gewebe (Maus, Schwein), in der konfokalen Laserscanning Mikroskopie - auch in Echtzeit - und in der Elektronenmikroskopie visualisiert.
ProjektleiterInnen:
Prof. Dr. Sven Hammerschmidt (Universität Greifswald)
Prof. Dr. Dr. h.c. Thomas C. Mettenleiter (FLI, Riems)
Projektmitarbeitende:
NachwuchsgruppenleiterInnen:
Dr. Nikolai Siemens (Universität Greifswald)
Dr. Theresa Schwaiger (FLI, Riems)
DoktorandInnen:
Surabhi Surabhi (Universität Greifswald)
Luca Zaeck (FLI, Riems)
Zur Analyse der Ubiquitinierung werden Transfektionsmodelle in immortalisierten und primären Lungenepithel-Zellkulturen etabliert, wobei überexprimierte Ubiquitin-Einheiten als "Target" für deren zelluläre Protein-Substrate dienen sollen. In weiterführenden Untersuchungen soll das ex vivo Ubiquitinom von infizierten Schweinen (Lunge, bronchoalveoläre Lavage, Immunzellen) analysiert und verglichen werden.
ProjektleiterInnen:
Prof. Dr. Dörte Becher (Universität Greifswald)
Prof. Dr. Ulrike Seifert (Universitätsmedizin Greifswald)
Projektmitarbeitende:
DoktorandInnen:
Thomas Sura (Universität Greifswald)
Vanessa Gering (Universitätsmedizin Greifswald)
