Die Mitarbeiter des Themengebietes Wirt-Pathogen Interaktionen charakterisieren auf Transkriptome- und Proteomeebene umfassend die Wechselwirkung von pathogenen Gram-positiven Bakterien wie z.B. S. aureus mit dem Menschen bzw. der Maus unter physiologisch relevanten Bedingungen, um die Pathophysiologie zu charakterisieren und neue Biomarkerkandidaten mit potentiellen diagnostischem und prognostischem Wert zu identifizieren. Zu diesem Zweck entwickeln und verbessern wir neue Methoden und Auswerteverfahren, um somit die Sensitivität und Spezifität von hochkomplexen Wirt-Pathogen Interaktionsdaten zu verbessern.
Verantwortliche Mitarbeiter
Wissenschaftliche Mitarbeiter
Dr. Kristin Surmann
Dr. Christian Hentschker
Dr. Petra Hildebrandt
Dr. Stephan Michalik
Doktoranden
Larissa Milena Busch
Denise Dittmar
Hannes Wolfgramm
Alexander Ganske
Technische Assistenz
Katrin Schoknecht
Spezifische Forschungsprojekte
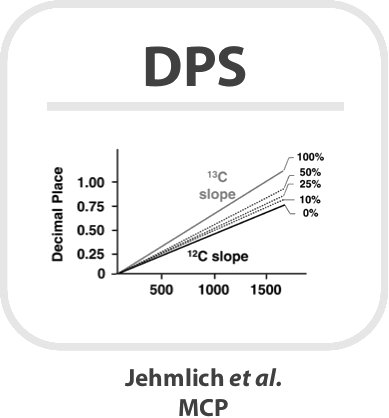
Die Isolierung von Bakterien aus Wirtsmaterial ist sehr komplex und führt in der Regel zu kleinen Mengen an Bakterien. Die geringe Anzahl an pathogenen Mikroorganismen erschweren jedoch die Identifizierung und Quantifizierung der bakterienspezifischen Proteine und erfordern hochspezialisierte Auswertemodule. In Zusammenarbeit mit unseren Partnern haben wir eine Vielzahl von solchen Massenspektrometrie-spezifischen Analyse- und Visualisierungswerkzeugen entwickelt bzw. bestehende Tools an die speziellen Gegebenheiten von Wirt-Pathogen Interaktionsassays angepasst, die nun eine umfassende Identifizierung und präzise Quantifizierung von Proteinen aus nur 105 Bakterienzellen ermöglichen. Einige dieser Werkzeuge sind rechts im Fenster abgebildet.
Gegenwärtig können 80 bis 90 Prozent der in einer Bakterienzelle exprimierten Proteine identifiziert und quantifiziert werden, wobei dafür gelfreie Massenspektrometrieverfahren erforderlich sind. Eine der wichtigsten Techniken der Abteilung ist die ESI-Massenspektrometrie, die mit Hilfe des BMBFs, der DFG, des Landes Mecklenburg-Vorpommerns und der EU gut in Greifswald etabliert werden konnte. Besonders Membranproteine, oberflächenassoziierte oder sezernierte Proteine sind von besonderer Bedeutung, da sie den ersten Kontakt mit dem Wirt darstellen. Eine Kombination verschiedener Quantifizierungsverfahren wie SILAC, metabolische Markierung oder labelfreie Techniken werden verwendet, um hochpräzise Proteine zu quantifizieren.
Bei klassischen Wirt-Pathogen Interaktionen sind die Bakterien meist in der Minderheit. So ist es immer eine Herausforderung, Proteine des Erregers auf Grund von Interferenz klar zu identifizieren und zu quantifizieren. In diesen Themengebiet wurde eine Vielzahl von Methoden entwickelt, um kleinste Mengen an Bakterien zu isolieren bzw. sortieren. Einige dieser Werkzeuge sind in den folgenden Abbildungen dargestellt.

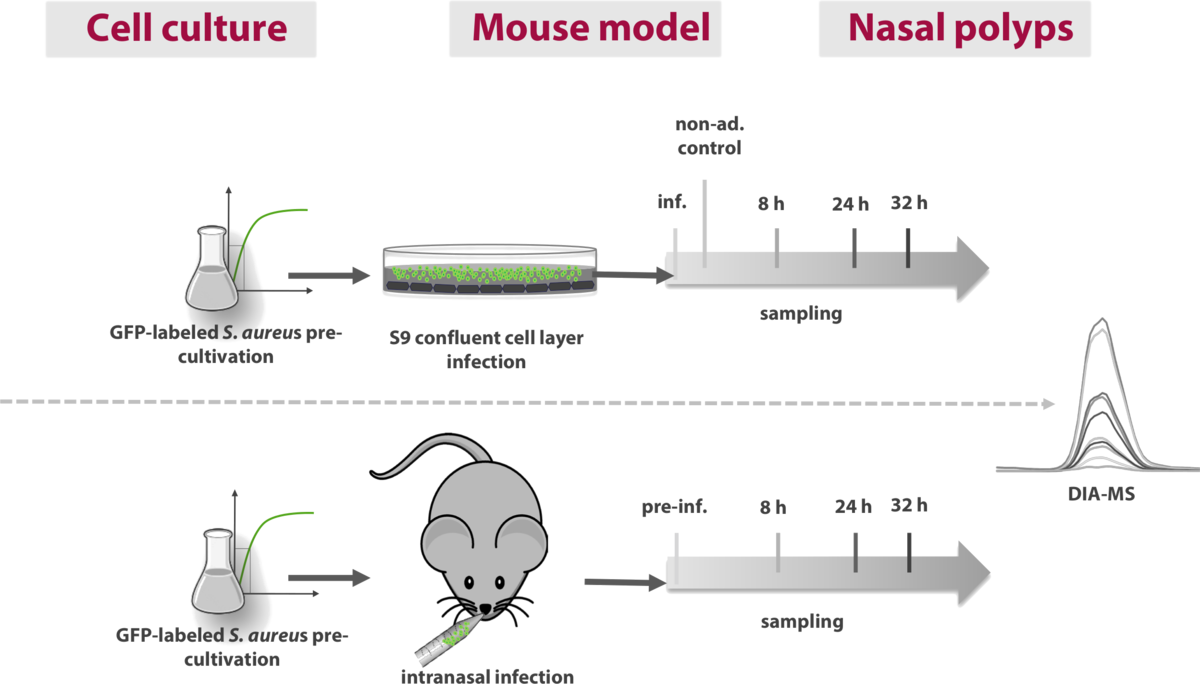
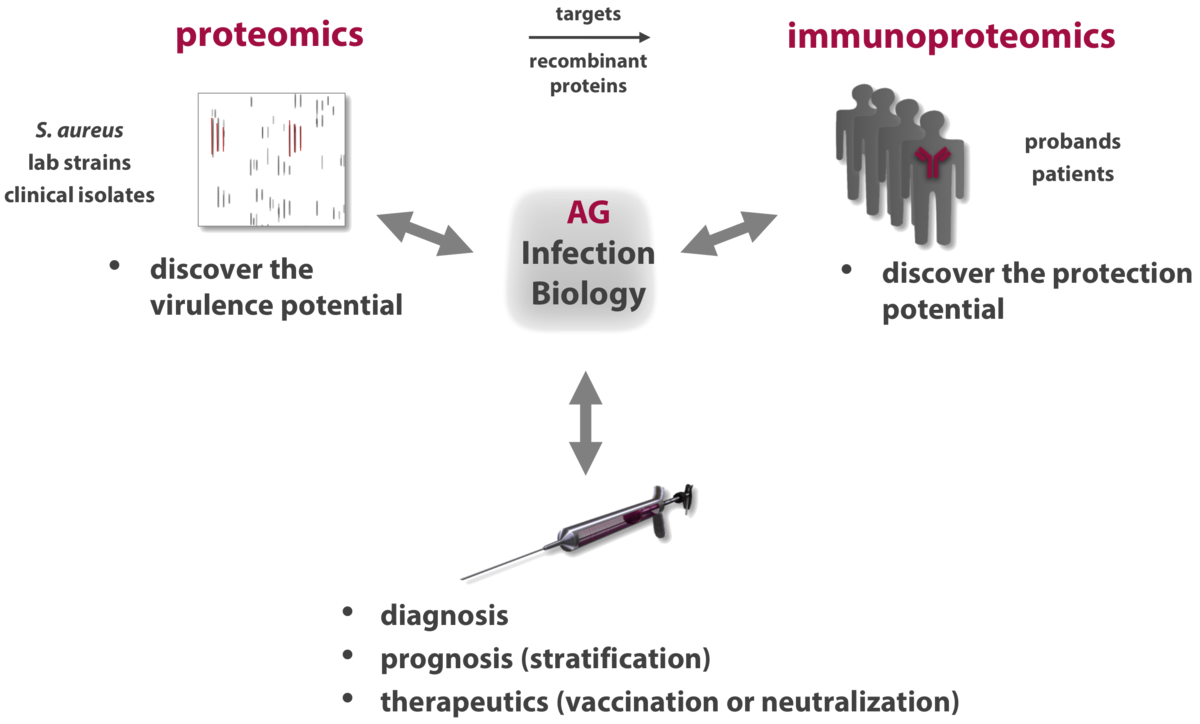
Um das Zusammenspiel von Wirt und seinem Pathogen zu verstehen, simulieren wir diese Interaktion in Zellkultur- oder Mausmodellen und untersuchen hierbei speziell die Proteome und Transkriptome beider Seiten. Weiterhin werden z.B. S. aureus-spezifische Nasenpolypen untersucht, um z.B. Virulenzfaktoren während der Kolonisation zu charakterisieren. Diese Untersuchungen sind für weitere immunoproteomische Studien von besonderem Interesse.
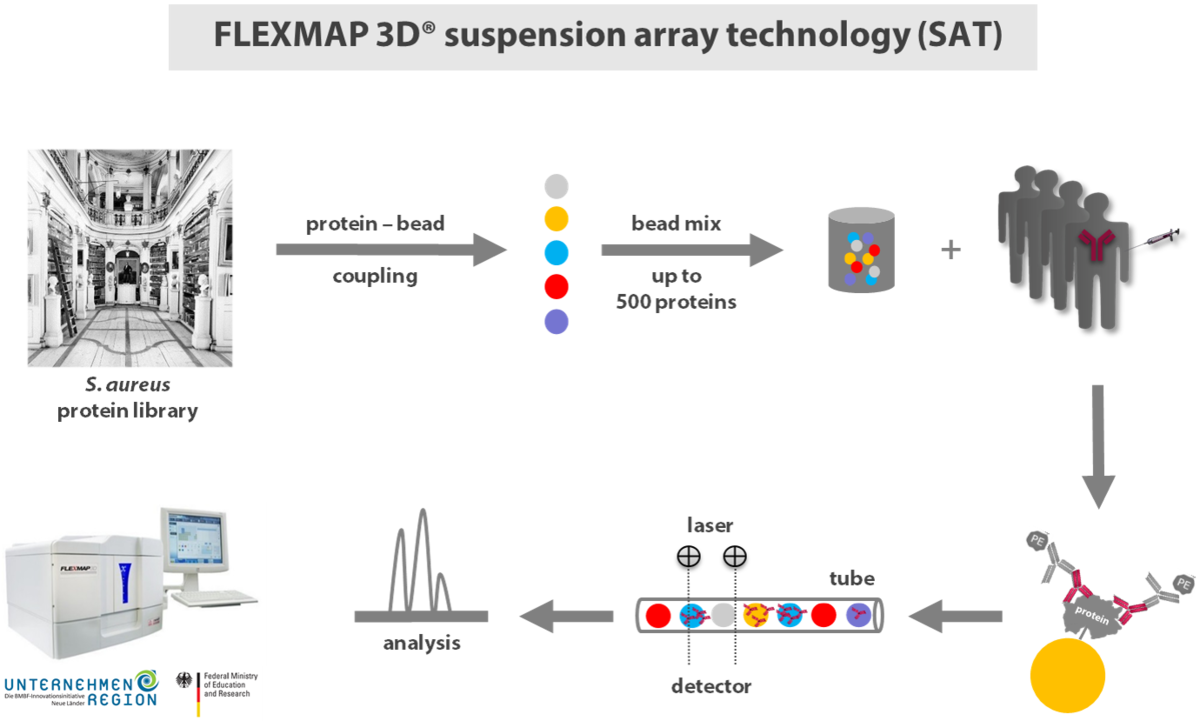
In Zusammenarbeit mit unseren Partnern charakterisieren wir die selektive Antikörperantwort gegen mikrobielle Virulenzfaktoren von Patienten mit infektionsbiologischen Hintergrund. Das Ziel ist es, eine Vielzahl von Antigentargets zu charakterisieren, die für eine verbesserte Diagnose oder Prognose geeignet sind. Auch sollen spezifische Moleküle herausgearbeitet werden, die zur Neutralisation von Virulenzfaktoren geeignet sind.
Förderinstitutionen
DFG | German Research Foundation
BMBF | Federal Ministry of Education and Research - InfectControl 2020
Federal State of Mecklenburg-Western Pomerania | KoInfekt
AiF | BMWi Germany| NanoPrep
Kooperationen
Barbara Bröker, Department of Immunology, Universitätsmedizin Greifswald
Sven Hammerschmidt, Department Genetic of Microorganisms, University of Greifswald
Michael Hecker, Katharina Riedel & Dörte Becher, Department of Microbial Physiology, University of Greifswald
Bernd Wollscheid, Institute of Molecular Systems Biology, ETH Zürich
Robert Moritz, Robert Moritz Group, Institute for Systems Biology
Hege Vangstein Aamot, The Department of Microbiology and Infection Control, Akershus University Hospital, Oslo, Norway
Yanina Lamberti, CONICET, National University of La Plata