Schwerpunkt des Themengebietes ist die Analyse physiologischer und pathologischer Proteomeprofile in humanen Geweben, Zellen, Bioflüssigkeiten bzw. zellulären und Tiermodellen. Im Mittelpunkt stehen dabei kardiosvaskuläre Erkrankungen, die im Rahmen des Deutschen Zentrums für Herz- und Kreislauf-Forschung untersucht werden. Darüber hinaus werden zusammen mit klinischen Forschungsgruppen u.a. onkologische und entwicklungsbiologische Fragestellungen untersucht.
Ziel ist es, Biomarker für die Diagnose von Erkrankungen bzw. die Prognose des Krankheitsverlaufes sowie Risikofaktoren zu identifizieren und pathophysiologische Prozesse besser zu verstehen. Dafür werden Methoden in der Proteomics adaptiert und entsprechende Auswertestrategien etabliert.
Verantwortliche Mitarbeiter
Wissenschaftliche Mitarbeiter
Dr. Sabine Ameling
Dr. Manuela Gesell Salazar
Dr. Vishnu Mukund Dhople
Technische Assistenz
Ulrike Lissner
Sophie Füsting
Spezifische Forschungsprojekte
Unser Verbund vereint die Expertisen aus der medizinischen Grundlagenforschung (Universitätsmedizin Greifswald), der funktionellen Proteinmodellierung (MIPS - Helmholtz-Institut für Pharmazeutische Forschung Saarland), der multi-Omics Netzwerkanalyse (Technische Universität München) und der klinischen Anwendung (Universitätsmedizin Greifswald).
Alternatives Spleißen (AS) ist ein biologischer Mechanismus durch den aus derselben DNA-Sequenz (Gen) verschiedene Transkripte (RNA-Kopien eines DNA-Abschnitts) und letztendlich verschiedene Proteine gebildet werden können. Das Gesamtziel von Sys_CARE ist es, die Bedeutung von AS für die Krankheitsentstehung und ihren Verlauf erstmals mit systemmedizinischen Ansätzen zu untersuchen und die hierfür nötigen bioinformatischen Methoden zu entwickeln.
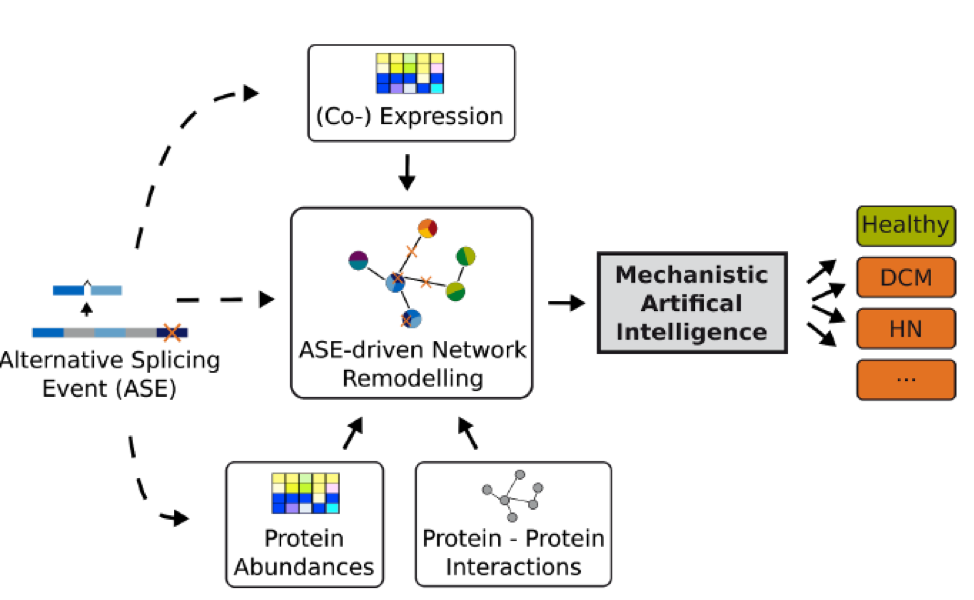
Zu diesem Zweck haben wir Zugriff auf einen großen bestehenden Datensatz aus molekularen und klinischen Daten, den wir im Zuge dieses Projektes komplementär ergänzen werden. Im Vordergrund steht die Entwicklung neuer Methoden, die es erlauben, molekularbiologisches Vorwissen in die Analyse der hochkomplexen Daten einzubringen und dadurch krankheitsrelevante Änderungen in AS aufzudecken, die mit bisherigen Methoden verborgen bleiben. Wichtige Teilziele des Projekts sind: Erstens, die Identifizierung von Transkripten, die bei Erkrankten signifikant häufiger oder seltener Vorkommen als bei gesunden Personen und es ggf. erlauben, Patienten mit dilatativer Kardiomyopathie (DCM) von Patienten mit hypertensiver Nephrosklerose (HN) sowie Kontrollprobanden zu unterscheiden. Bei DCM handelt es sich um eine krankhafte Erweiterung des Herzmuskels mit Funktionseinbußen, während HN eine entzündungsfreie Nierenerkrankung infolge von Bluthochdruck ist. Zweitens, die Aufklärung der Rolle von miRNAs (kleinen regulatorischen Transkripten) zur Regulation von AS, sowie die Untersuchung des therapeutischen Potentials von miRNAs um zielgerichtet in krankhafte AS-Prozesse einzugreifen.
Im Zuge der digitalen Revolution hat die Nutzung und Verarbeitung personenbezogener Daten besondere Bedeutung für die allgemeine Wertschöpfungskette erlangt. Dies erfordert einerseits einen reibungslosen Datenaustausch mit hoher Datenqualität und andererseits einen entsprechenden Datenschutz. In TreuMed sollen am Beispiel der „verteilten künstlichen Intelligenz“ in der Medizin Datentreuhandmodelle und entsprechende Geschäftskonzepte entwickelt und erprobt werden, die den Datenschutz gewährleisten und gleichzeitig Anreize für Innovationsförderung schaffen. Mit der „verteilten künstlichen Intelligenz“ in der Medizin stellen wir eine technische Lösung vor, mit deren Hilfe sich Patientendaten unter Wahrung datenschutzrechtlicher Anforderungen innovationsförderlich nutzen lassen. Dieser technische Ansatz sorgt für einen umfassenden Datenschutz „by design“: Patientendatensätze müssen das Krankenhaus zu keinem Zeitpunkt verlassen. Nur aggregierte, synthetische Daten (Modellparameter) werden für Forschungszwecke weiterverarbeitet.

Wie sich die „verteilte künstliche Intelligenz“ mit Hilfe von Datentreuhändern rechtssicher implementieren lässt und welche Geschäftsmodelle sich dafür eignen, möchten wir mit dem hier vorgeschlagenen Forschungsverbundprojekt aufzeigen. Wir definieren den Rechtsrahmen für Datentreuhandmodelle und entwickeln passende Geschäftsmodelle für dieses Anwendungsbeispiel. Den Kern dieser Modelle bildet ein Ampelsystem für Datentreuhänder mit drei möglichen Privacy-Ebenen und entsprechendem Schutzvorkehrungslevel flankiert durch Zertifizierungspflichten und Haftungskonzepte für Restrisiken. Die Erprobung der vorgestellten Modelle erfolgt durch zwei medizinische Anwendungen der „verteilten künstlichen Intelligenz“ im Bereich der molekularen Epidemiologie und der Biomarkerforschung am Beispiel (identifizierender) Genomik-, Transkriptomik- und Bild-Daten.
Im Rahmen des Deutschen Zentrums für Herz-und Kreislauferkrankungen werden Protein-, mRNA- und miRNA-Analysen von Patientenproben durchgeführt, die zu einem besseren Verständnis von Pathomechanismen kardiovaskulärer Erkrankungen bzw. der molekularen Veränderungen bei Therapie beitragen sollen. Der Fokus liegt dabei auf der Dilatativen Kardiomyopathie (DCM). Diese Herzmuskelerkrankung ist durch die Abnahme der kontraktilen Funktion des Myokards und durch eine Dilatation des rechten und linken Ventrikels gekennzeichnet.
Insgesamt wächst jedoch die Bedeutung der Herzinsuffizienz (HI- Herzschwäche) mit ihren ätiologisch sehr verschiedenen Ursachen und damit die Notwendigkeit für eine bessere Diagnostik und die Entwicklung verbesserter Therapieansätze. Voraussetzung dafür ist die Untersuchung von Proben in bestehenden und neu etablierten Patientenkohorten zum besseren Verständnis der molekularen Adaptionen in der HI.
Ziel der Arbeiten ist es daher, durch ein Screening von Bioproben Unterschiede im RNA- und Proteinmuster sowohl im Gewebe als auch in Bioflüssigkeiten von HI-Patienten und Kontrollpatienten ohne eingeschränkte Herzfunktion zu erkennen. Mit diagnostischen molekularen Signaturen sollen Patienten-Subgruppen klassifiziert und anhand der Prädiktion des Krankheitsverlaufes Therapieanpassungen ermöglicht werden.
Kooperationspartner werden bei der molekularen Charakterisierung von Zell- und Tiermodellen für kardiovaskuläre Erkrankungen durch die Erstellung von Protein- und Phosphopeptidprofilen unterstützt.
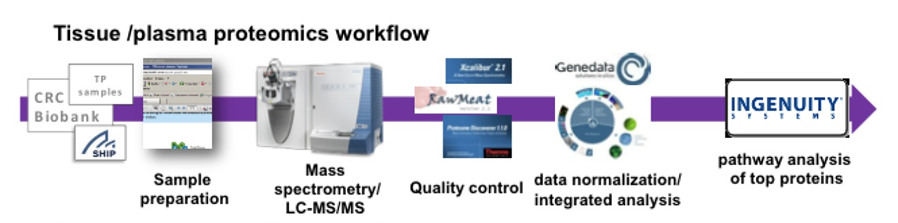
Die Entwicklung individueller Therapieoptionen gewinnt zunehmend an Bedeutung. Die OMICS-Technologien eröffnen dabei neue Möglichkeiten für die Identifikation neuer Biomarkerkandidaten zur Unterstützung der Diagnose und Prognose von Erkrankungen und Ableitung individueller Therapieansätze. Im Fokus unserer Arbeiten stehen Profilingstudien im Blut von Probanden der Bevölkerungsstudie SHIP bzw. von Patienten mit kardiovaskulären Erkrankungen. Neben den dafür notwendigen Methoden für das Profiling mehrerer hundert Proben für miRNA mittels PCR und für Proteine auf Basis neuester massenspektrometrischer Technologien wurden Arbeitsabläufe für die Standardisierung, die Qualitätskontrolle sowie die individuelle, aber auch integrierte Auswertung von Datensätzen sowie deren Interpretation erarbeitet und können nun für die Analyse der verschiedensten Phänotypen angewendet werden. Komplettiert wird das Analyseportfolio durch die Anwendung der Methoden auf Untersuchungen an Speichel und Urin.
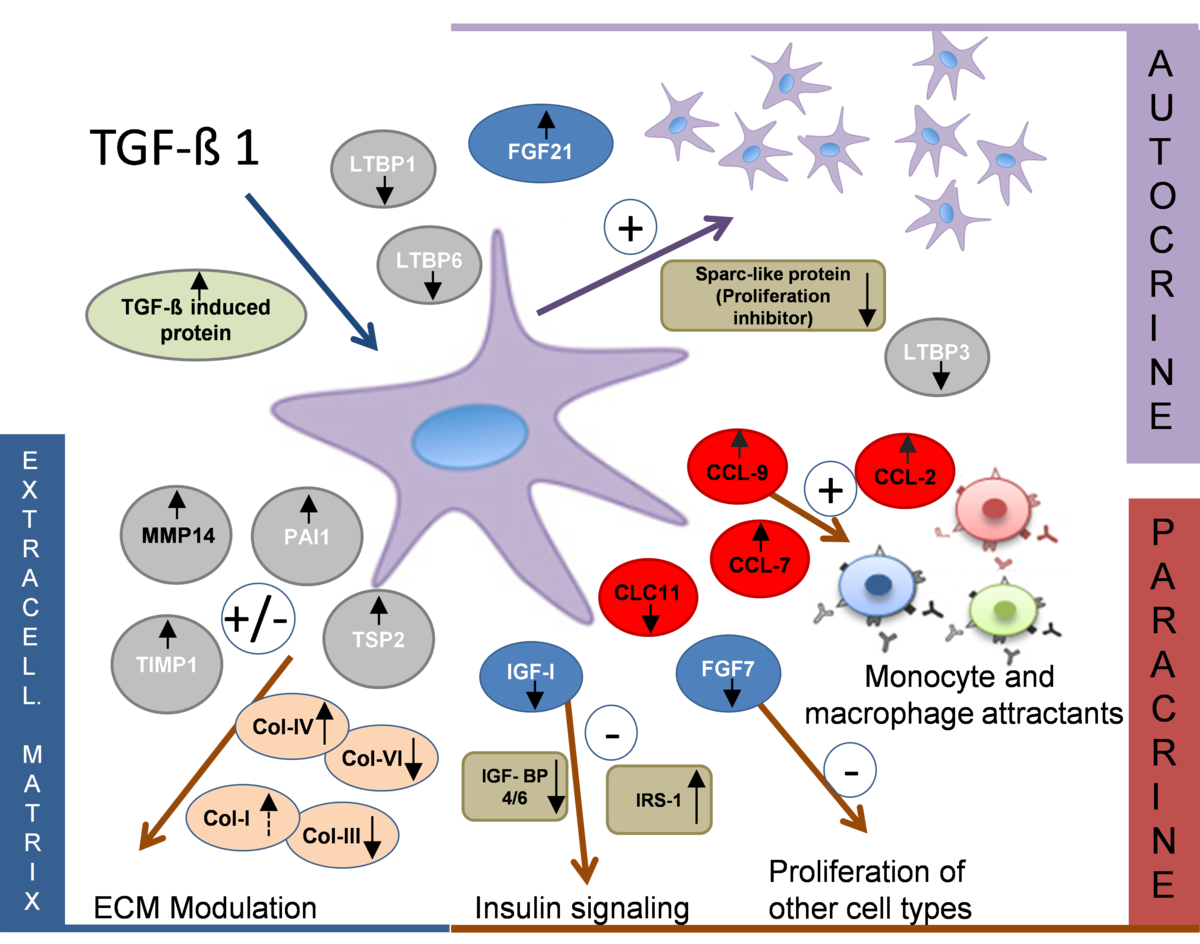
Zellen sekretieren Proteine, die eine wichtige Rolle bei der Erhaltung der Zellintegrität und –stabilität, Mobilisierung und Differenzierung spielen. Solche Proteine können als autokrine (Wirkung auf Zellen gleichen Typs) oder parakrine (Wirkung auf andere Zellarten) Faktoren wirken und initiieren indirekt eine Zellantwort auf externe Stimuli. Die Analyse sekretierter Proteine auch aus Zellkulturen erfordert besondere Arbeitsabläufe, die die geringe Menge dieser Proteinspezies, das Vorliegen in niedrigen Konzentrationen sowie die Interferenz mit Mediumbestandteilen berücksichtigen. In der Arbeitsgruppe wurden adaptierte Zellkulturprotokolle und Proteinanreicherungsstrategien etabliert, die eine umfassende massenspektrometrische Analyse von Sekretomen auch aus geringen Zellzahlen wie sie u.a. bei Stammzellen vorliegen ermöglichen.
Förderinstitutionen
DZHK | German Centre for Cardiovascular Research
BMBF | Federal Ministry of Education and Research
DFG | German Research Foundation
Kooperationen
Stephan Felix, Department for Internal Medicine B, University Medicine Greifswald
Jens Fielitz, Department for Internal Medicine B, University Medicine Greifswald
Thomas Kocher, Department of Restorative Dentistry, Periodontology, Endodontology, Preventive and Pediatric Dentistry, University Medicine Greifswald
Nicole Endlich, Department of Anatomy and Cell Biology, University Medicine Greifswald
Matthias Heckmann, Department of Neonatology and Pediatric Intensive Care, University Medicine Greifswald
Karin Klingel, Department of Molecular Pathology, University Hospital Tübingen, Germany
Dirk Westermann, University Heart Center, University Hospital Hamburg
Cristina Iuga, Department of Pharmaceutical Analysis, University of Medicine and Pharmacy "Iuliu Hatieganu", Cluj, Romania
Verena Stangl & Mario Lorenz, AG Lorenz/ Stangl, Charité Berlin